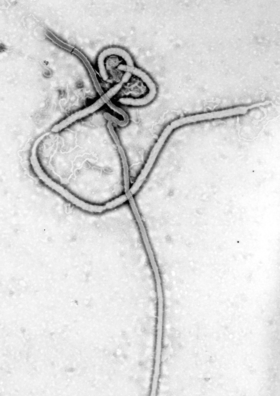
Vírus Ebola
Vírus Ebola (português brasileiro) ou Vírus Ébola (português europeu)[1] (EBOV, anteriormente designado Ebolavírus Zaire) é o único membro da espécie Zaire ebolavírus e o mais perigoso dos cinco vírus conhecidos dentro do gênero Ebolavírus.[2] Quatro dos cinco ebolavírus conhecidos causam febre e uma hemorragia grave e muitas vezes fatal em seres humanos e outros mamíferos, patologia conhecida febre hemorrágica Ebola. O vírus e suas espécies foram originalmente chamados de Zaire (hoje República Democrática do Congo), o país onde foi descrito pela primeira vez[2] e quando houve a primeira suspeita de ser uma nova "estirpe" do vírus de Marburg, intimamente relacionado;[3][4][4] o vírus (mas não a sua espécie) foi renomeado para "vírus Ebola", em 2010, para evitar confusão. A espécie é uma espécie de táxon virológico incluído no gênero Ebolavirus, família Filoviridae (cujos membros são chamados de Filovírus), ordem Mononegavirales.[2] Acredita-se que seu reservatório natural sejam morcegos, particularmente morcegos frugívoros, e é transmitida principalmente entre os seres humanos e de animais para humanos, através de fluidos corporais.
| Vírus Ébola | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Classificação científica | |||||||||
| |||||||||
O vírus Ebola causou a maioria das mortes humanas por doença por vírus Ébola (DVE) e foi a causa da epidemia de 2013-2016 na África Ocidental,[5] que resultou em pelo menos 28.646 casos suspeitos e 11.323 mortes confirmadas.[6]
O genoma do EBOV é de aproximadamente 19.000 nucleotídeos. Ele codifica sete proteínas estruturais:. nucleoproteína (NP), polimerase cofator (VP35), (VP40), GP, ativador de transcrição (VP30), VP24 e ARN-polimerase (L).[7]
Devido à sua alta taxa de mortalidade, o EBOV também é listado como um agente de seleção. Foi classificado como um patógeno de Risco 4 pela Organização Mundial de Saúde (o que exige Nível de Biossegurança 4 contenção equivalente); um patógeno de Categoria A pelos Institutos Nacionais da Saúde/Instituto Nacional de Alergia e Doenças Infecciosas; um agente de bioterrorismo de Categoria A pelo Centro de Controle e Prevenção de Doenças; além de ser listado pelo Grupo Austrália como um agente biológico que deve ser controlado.
Estrutura editar
O EBOV carrega um genoma de RNA de sentido negativo em vírions que são cilíndricos/tubulares e contêm componentes de envelope viral, matriz e nucleocapsídeo. O vírus geralmente possui aproximadamente 80 nm de diâmetro e 970 nm de comprimento e possui uma glicoproteína (GP) codificada pelo vírus que se projeta como espículas de 7 a 10 nm de comprimento a partir de sua superfície de bicamada lipídica.[8] O envelope viral do vírion é derivado por brotamento dos domínios da membrana da célula hospedeira nos quais as espículas de GP foram inseridas durante sua biossíntese. As moléculas individuais de GP aparecem com espaçamentos de cerca de 10 nm. As proteínas virais VP40 e VP24 estão localizadas entre o envelope e o nucleocapsídeo (veja a seguir), no espaço da matriz.[9] No centro da estrutura do vírion está o nucleocapsídeo, que é composto por uma série de proteínas virais ligadas a um RNA de sentido negativo linear de 18–19 kb, sem poliadenilação, de 3′ ou 5′ (veja a seguir); o RNA é enrolado helicoidalmente e complexado com as proteínas NP, VP35, VP30 e L. Essa hélice tem um diâmetro de 80 nm.[10][10][11]
A forma geral dos vírions após a purificação e a visualização (por exemplo, por ultracentrifugação e microscopia eletrônica, respectivamente) varia consideravelmente; cilindros simples são muito menos prevalentes do que estruturas em direção inversa, ramificações e loops (por exemplo, formas de cajado U, de parafuso olhal 9 ou outras aparências circulares/enroladas), cuja origem pode estar nas técnicas laboratoriais aplicadas.[12][13] A estrutura "filiforme" característica é, no entanto, uma característica morfológica mais geral dos filovírus (juntamente com seu envelope viral com glicoproteína, nucleocapsídeo de RNA, etc.).[12]
Genoma editar
Cada vírion contém uma molécula de RNA linear, de fita simples e sentido negativo, com 18.959 a 18.961 nucleotídeos de comprimento.[14] A extremidade 3′ não é poliadenilada e a extremidade 5′ não possui cap. Esse genoma viral codifica sete proteínas estruturais e uma proteína não estrutural. A ordem dos genes é 3′ - leader - NP - VP35 - VP40 - GP/sGP - VP30 - VP24 - L - trailer - 5′; sendo que o leader e o trailer são regiões não transcritas, que carregam sinais importantes para controlar a transcrição, a replicação e o empacotamento dos genomas virais em novos vírions. As seções dos genes NP, VP35 e L dos filovírus foram identificadas como endógenas nos genomas de vários grupos de pequenos mamíferos.[15][16][17]
Descobriu-se que 472 nucleotídeos da extremidade 3' e 731 nucleotídeos da extremidade 5' são suficientes para a replicação de um "minigenoma" viral, embora não sejam suficientes para a infecção.[12] O sequenciamento do vírus de 78 pacientes com doença confirmada pelo vírus Ebola, representando mais de 70% dos casos diagnosticados em Serra Leoa do final de maio a meados de junho de 2014,[18][19] forneceu evidências de que o surto de 2014 não estava mais sendo alimentado por novos contatos com seu seu reservatório natural. Usando a tecnologia de sequenciamento de terceira geração, os pesquisadores conseguiram sequenciar amostras em apenas 48 horas.[20] Como outros vírus de RNA,[18] o vírus Ebola sofre mutação rapidamente, tanto dentro de uma pessoa durante a progressão da doença quanto no reservatório entre a população humana local.[19] A taxa de mutação observada de 2,0 x 10−3 substituições por local por ano é tão rápida quanto a da gripe sazonal.[21]
| Símbolo | Nome | UniProt | Função |
|---|---|---|---|
| NP | Nucleoproteína | P18272 | Envolve o genoma para proteção contra nucleases e imunidade inata. |
| VP35 | Polimerase cofator VP35 | Q05127 | Polimerase cofator; suprime a imunidade inata ao se ligar ao RNA. |
| VP40 | Matriz proteica VP40 | Q05128 | Matriz. |
| GP | Envelope de glicoproteína | Q05320 | Clivado pela furina do hospedeiro em GP1/2 para formar um envelope com espículas. Também libera GP como chamariz. |
| sGP | Glicoproteína segregada | P60170 | Compartilha a fase de leitura aberta (ORF) com a GP. Clivada pela furina do hospedeiro em sGP (anti-inflamatória) e peptídeo delta (viroporina). |
| ssGP | Glicoproteína viral | Q9YMG2 | Compartilha a ORF com a GP; criada por edição de mRNA. Função desconhecida. |
| VP30 | Proteína dedo de zinco VP30 hexamérica | Q05323 | Ativador transcricional. |
| VP24 | Proteína VP24 associada à membrana | Q05322 | Bloqueia a sinalização de IFN-alfa/beta e gama. |
| L | RNA polimerase (L) dependente de RNA | Q05318 | Replicase de RNA. |
Entrada editar
Há dois candidatos a proteínas de entrada na célula hospedeira. A primeira é uma proteína transportadora de colesterol, a C1 de Niemann-Pick (NPC1) codificada pelo hospedeiro, que parece ser essencial para a entrada dos vírions do Ebola na célula hospedeira e para sua replicação final.[22][23] Em um estudo, camundongos com uma cópia do gene NPC1 inativado apresentaram uma taxa de sobrevivência de 80% quinze dias após a exposição ao vírus Ebola adaptado para camundongos, enquanto apenas 10% dos camundongos não modificados sobreviveram por esse tempo.[22] Em outro estudo, foi demonstrado que micromoléculas inibem a infecção pelo vírus Ebola ao impedir que a glicoproteína (GP) do envelope viral se ligue à NPC1.[23][24] Portanto, a NPC1 demonstrou ser essencial para a entrada desse filovírus, pois medeia a infecção ligando-se diretamente à GP viral.[23]
Quando as células de indivíduos com Doença de Niemann-Pick (que não tinham o transportador NPC1) foram expostas ao vírus Ebola em laboratório, as células sobreviveram e pareciam impermeáveis ao vírus, indicando ainda que o Ebola depende do NPC1 para entrar nas células.[22] Mutações no gene NPC1 em humanos foram conjecturadas como um possível modo de tornar alguns indivíduos resistentes a essa doença viral mortal. Os mesmos estudos descreveram resultados semelhantes em relação à função do NPC1 para o vírus de Marburg, um filovírus semelhante.[22] Um outro estudo também apresentou evidências de que o gene NPC1 é o receptor crítico que medeia a infecção pelo Ebola por meio de sua ligação direta à GP viral e que é o segundo domínio "lisossômico" do NPC1 que medeia essa ligação.[25]
O segundo candidato é o TIM-1 (também conhecido como HAVCR1).[26] Foi demonstrado que o TIM-1 se liga ao domínio de ligação do receptor da glicoproteína do EBOV para aumentar a receptividade das células Vero. O bloqueio do seu efeito com siRNA impediu a infecção das células Vero. O TIM1 é expresso em tecidos conhecidos por serem seriamente afetados pela lise do EBOV (traqueia, córnea e conjuntiva). Um anticorpo monoclonal contra o domínio IgV do TIM-1, ARD5, bloqueou a ligação e a infecção pelo EBOV. Juntos, esses estudos sugerem que o NPC1 e o TIM-1 podem ser alvos terapêuticos em potencial para um medicamento antiviral contra o Ebola e como base para um ensaio de diagnóstico de campo rápido.
Replicação editar
Por serem acelulares, os vírus como o Ebola não se replicam por meio de nenhum tipo de divisão celular; em vez disso, eles usam uma combinação de enzimas codificadas pelo hospedeiro e pelo vírus, juntamente com estruturas da célula hospedeira, para produzir várias cópias de si mesmos. Em seguida, essas cópias se automontam em estruturas macromoleculares virais na célula hospedeira.[27] O vírus conclui um conjunto de etapas ao infectar cada célula individual. O vírus começa seu ataque ligando-se aos receptores do hospedeiro por meio do peplômero da superfície da glicoproteína (GP) e é endocitado em macropinossomos na célula hospedeira.[28] Para penetrar na célula, a membrana viral se funde com a membrana da vesícula e o nucleocapsídeo é liberado no citoplasma. O ssRNA genômico de sentido negativo encapsidado é usado como modelo para a síntese (3'-5') de mRNAs monocistrônicos poliadenilados e, usando os ribossomos da célula hospedeira, moléculas de tRNA, etc., o mRNA é traduzido em proteínas virais individuais.[29][30][31]
Essas proteínas virais são processadas: um precursor de glicoproteína (GP0) é clivado em GP1 e GP2, que são então fortemente glicosiladas usando enzimas e substratos celulares. Essas duas moléculas se reúnem, primeiro em heterodímeros e, depois, em trímeros para formar os peplômeros de superfície. O precursor da glicoproteína segregada secretada (sGP) é clivado em sGP e polipeptídeo (polipeptídeo), ambos liberados da célula. À medida que os níveis de proteína viral aumentam, ocorre uma mudança da tradução para a replicação. Usando o RNA genômico de sentido negativo como modelo, um +ssRNA complementar é sintetizado; esse é então usado como modelo para a síntese do novo (-)ssRNA genômico, que é rapidamente encapsidado. Os nucleocapsídeos recém-formados e as proteínas do envelope se associam à membrana plasmática da célula hospedeira e ocorre a excreção viral, destruindo a célula.
Ecologia editar
O vírus Ebola é um patógeno zoonótico. Foi relatado que os hospedeiros intermediários são "várias espécies de morcegos frugívoros...em toda a África central e subsariana". A evidência de infecção em morcegos foi detectada por meios moleculares e sorológicos. No entanto, o vírus não foi isolado em morcegos.[32][33] Os hospedeiros finais são humanos e grandes primatas, infectados por contato com morcegos ou por outros hospedeiros finais. Foi relatado que porcos nas Filipinas foram infectados com o vírus Reston, portanto, podem existir outros hospedeiros intermediários ou amplificadores.[33] Os surtos do vírus Ebola tendem a ocorrer quando as temperaturas são mais baixas e a umidade é mais alta do que o normal na África.[34] Mesmo depois que uma pessoa se recupera da fase aguda da doença, o vírus Ebola sobrevive por meses em determinados órgãos, como olhos e testículos.[35]
Doença por vírus Ebola editar
O Zaire ebolavírus é um dos quatro vírus Ebola conhecidos por causar doenças em humanos. Ele tem a maior taxa de letalidade entre esses vírus, com uma média de 83% desde os primeiros surtos em 1976, embora uma taxa de letalidade de até 90% tenha sido registrada em um surto na República do Congo entre dezembro de 2002 e abril de 2003. Também houve mais surtos do Zaire ebolavírus do que de qualquer outro vírus Ebola. O primeiro surto ocorreu em 26 de agosto de 1976 em Yambuku.[36] O primeiro caso registrado foi o de Mabalo Lokela, um professor de 44 anos. Os sintomas se assemelhavam aos da malária, e os pacientes subsequentes receberam quinino. A transmissão foi atribuída à reutilização de agulhas não esterilizadas e ao contato pessoal próximo, fluidos corporais e locais onde a pessoa tocou. Durante o surto de Ebola de 1976 no Zaire, Ngoy Mushola viajou de Bumba para Yambuku, onde registrou a primeira descrição clínica da doença em seu diário:
"A doença é caracterizada por uma temperatura elevada de cerca de 39°C, hematemese, diarreia com sangue, dor abdominal retroesternal, prostração com articulações "pesadas" e morte de evolução rápida após uma média de três dias."[37]
Desde a primeira descrição clínica registrada da doença em 1976 no Zaire, o surto de Ebola, que começou em março de 2014, atingiu proporções epidêmicas e matou mais de 8.000 pessoas em janeiro de 2015. Esse surto foi centrado na África Ocidental, uma área que não havia sido afetada anteriormente pela doença. O número de vítimas foi particularmente grave em três países: Guiné, Libéria e Serra Leoa. Alguns casos também foram relatados em países fora da África Ocidental, todos relacionados a viajantes internacionais que foram expostos nas regiões mais afetadas e posteriormente apresentaram sintomas da febre Ebola após chegarem a seus destinos.[38]
A gravidade da doença em humanos varia amplamente, desde a rápida fatalidade até a doença leve ou até mesmo a resposta assintomática.[39] Estudos de surtos no final do século XX não conseguiram encontrar uma correlação entre a gravidade da doença e a natureza genética do vírus. Portanto, suspeitou-se que a variabilidade na gravidade da doença estivesse correlacionada com as diferenças genéticas das vítimas. Tem sido difícil estudar isso em modelos animais que respondem ao vírus com febre hemorrágica de maneira semelhante à dos seres humanos, porque os modelos típicos de camundongos não respondem dessa forma e o grande número necessário de cobaias apropriadas não está facilmente disponível. No final de outubro de 2014, uma publicação relatou um estudo da resposta a uma cepa adaptada de camundongo do Zaire ebolavírus apresentada por uma população geneticamente diversa de camundongos que foi criada para ter uma gama de respostas ao vírus que inclui a fatalidade da febre hemorrágica.[40]
Vacina editar
Em dezembro de 2016, um estudo constatou que a vacina VSV-EBOV era 70-100% eficaz contra o vírus do Zaire (não o do Sudão), tornando-a a primeira vacina contra a doença.[41][42] A VSV-EBOV foi aprovada pela Administração de Alimentos e Medicamentos dos EUA (FDA) em dezembro de 2019.[43]
História e nomenclatura editar
O vírus Ebola foi identificado pela primeira vez como uma possível nova "cepa" do vírus de Marburg em 1976.[3][4][44] O Comitê Internacional de Taxonomia de Vírus (ICTV) identifica o vírus Ebola como a espécie Zaire ebolavirus, que faz parte do gênero Ebolavirus, família Filoviridae, ordem Mononegavirales. O nome "vírus Ebola" é derivado do rio Ebola - um rio que, a princípio, acreditava-se estar próximo à área da República Democrática do Congo, anteriormente chamada de Zaire, onde ocorreu o surto do vírus Ebola do Zaire em 1976 - e do sufixo taxonômico virus.[2][3][4][45]
Em 1998, o nome do vírus foi alterado para "Zaire Ebola virus"[46][47] e, em 2002, para a espécie Zaire ebolavirus.[48][49] Entretanto, a maioria dos artigos científicos continuou a se referir ao "vírus Ebola" ou usou os termos "vírus Ebola" e "Zaire ebolavirus" em paralelo. Consequentemente, em 2010, um grupo de pesquisadores recomendou que o nome "vírus Ebola" fosse adotado para uma subclassificação dentro da espécie Zaire ebolavirus, com a abreviação EBOV correspondente.[2] As abreviações anteriores para o vírus eram EBOV-Z (para "vírus Ebola Zaire") e ZEBOV (para "vírus Ebola Zaire" ou "Zaire ebolavirus"). Em 2011, o ICTV rejeitou explicitamente uma proposta (2010.010bV) para reconhecer esse nome, pois o ICTV não designa nomes para subtipos, variantes, cepas ou outros agrupamentos em nível de subespécie.[50] Atualmente, o ICTV não reconhece oficialmente o "vírus Ebola" como uma classificação taxonômica, mas continua a usar e recomendar apenas a designação de espécie Zaire ebolavirus.[51] O tipo nomenclatural do vírus Ebola, variante Mayinga (EBOV/May), foi nomeado em homenagem a Mayinga N'Seka, uma enfermeira que morreu durante o surto de 1976 no Zaire.[2][52][53]
O nome Zaire ebolavirus é derivado de Zaire e do sufixo taxonômico ebolavirus (que denota uma espécie de ebolavírus e se refere ao rio Ebola).[2] De acordo com as regras de nomenclatura de táxons estabelecidas pelo Comitê Internacional de Taxonomia de Vírus (ICTV), o nome Zaire ebolavirus deve ser sempre maiúsculo, em itálico e precedido pela palavra "espécie". Os nomes de seus membros (Zaire ebolavirus) devem ser capitalizados, não devem estar em itálico e devem ser usados sem artigos.[2]
Critérios de inclusão de vírus editar
Um vírus do gênero Ebolavirus é um membro da espécie Zaire ebolavirus se:[2]
- For endêmico na República Democrática do Congo, no Gabão ou na República do Congo;
- tiver um genoma com duas ou três sobreposições de genes (VP35/VP40, GP/VP30, VP24/L);
- tiver um genoma que difere do vírus tipo EBOV/May em menos de 30%.
Evolução editar
A diversidade genética do Zaire ebolavirus divergiu de seus ancestrais entre 1960 e 1976.[54] A diversidade genética do ebolavírus permaneceu constante antes de 1900.[54][55] Então, por volta da década de 1960, provavelmente devido à mudança climática ou às atividades humanas, a diversidade genética do vírus caiu rapidamente e a maioria das linhagens foi extinta.[55] À medida que o número de hospedeiros suscetíveis diminui, o mesmo acontece com o tamanho efetivo da população e sua diversidade genética. Esse efeito de funil genético tem implicações para a capacidade da espécie de causar a doença por vírus Ebola em hospedeiros humanos.
Um evento de recombinação entre as linhagens do Zaire ebolavirus provavelmente ocorreu entre 1996 e 2001 em macacos selvagens, dando origem a vírus descendentes recombinantes.[56] Esses vírus recombinantes foram supostamente responsáveis por uma série de surtos entre humanos na África Central em 2001-2003.[56]
O Zaire ebolavirus - variante Makona causou o surto de 2014 na África Ocidental.[57] O surto foi caracterizado pela mais longa instância de transmissão de humano para humano da espécie viral.[57] Pressões para se adaptar ao hospedeiro humano foram observadas nesse momento, no entanto, nenhuma mudança fenotípica no vírus (como aumento da transmissão, aumento da evasão imunológica pelo vírus) foi observada.
Ver também editar
Referências
- ↑ Correia, Paulo (Verão de 2022). «Os poxvírus e o vírus da perda de domínios» (PDF). a folha — Boletim da língua portuguesa nas instituições europeias. Consultado em 2 de fevereiro de 2023
- ↑ a b c d e f g h i Kuhn, Jens H.; Becker, Stephan; Ebihara, Hideki; Geisbert, Thomas W.; Johnson, Karl M.; Kawaoka, Yoshihiro; Lipkin, W. Ian; Negredo, Ana I; et al. (2010). «Proposal for a revised taxonomy of the family Filoviridae: Classification, names of taxa and viruses, and virus abbreviations». Archives of Virology. 155 (12): 2083–103. PMC 3074192 . PMID 21046175. doi:10.1007/s00705-010-0814-x
- ↑ a b c Pattyn, S.; Jacob, W.; van der Groen, G.; Piot, P.; Courteille, G. (1977). «Isolation of Marburg-like virus from a case of haemorrhagic fever in Zaire». Lancet (em inglês). 309 (8011): 573–4. PMID 65663. doi:10.1016/s0140-6736(77)92002-5
- ↑ a b c d Bowen, E. T. W.; Lloyd, G.; Harris, W. J.; Platt, G. S.; Baskerville, A.; Vella, E. E. (1977). «Viral haemorrhagic fever in southern Sudan and northern Zaire. Preliminary studies on the aetiological agent». Lancet (em inglês). 309 (8011): 571–3. PMID 65662. doi:10.1016/s0140-6736(77)92001-3 Erro de citação: Código
<ref>inválido; o nome "Bowen1977" é definido mais de uma vez com conteúdos diferentes - ↑ Na, Woonsung; Park, Nanuri; Yeom, Minju; Song, Daesub (4 de dezembro de 2016). «Ebola outbreak in Western Africa 2014: what is going on with Ebola virus?». Clinical and Experimental Vaccine Research (em inglês). 4 (1): 17–22. ISSN 2287-3651. PMC 4313106 . PMID 25648530. doi:10.7774/cevr.2015.4.1.17
- ↑ «Ebola virus disease outbreak». World Health Organization (em inglês). Consultado em 4 de dezembro de 2016
- ↑ Nanbo, Asuka; Watanabe, Shinji; Halfmann, Peter; Kawaoka, Yoshihiro (4 de fevereiro de 2013). «The spatio-temporal distribution dynamics of Ebola virus proteins and RNA in infected cells». Nature (em inglês). doi:10.1038/srep01206
- ↑ Klenk, H.-D.; Feldmann, H., eds. (2004). Ebola and Marburg Viruses – Molecular and Cellular Biology (em inglês). Wymondham, Norfolk, UK: Horizon Bioscience. p. 28. ISBN 978-0-9545232-3-7
- ↑ Feldmann, H. K. (1993). «Molecular biology and evolution of filoviruses». Unconventional Agents and Unclassified Viruses. Archives of Virology. Supplementum. Col: Archives of Virology (em inglês). 7. [S.l.: s.n.] pp. 81–100. ISBN 978-3211824801. ISSN 0939-1983. PMID 8219816. doi:10.1007/978-3-7091-9300-6_8
- ↑ a b Lee, Jeffrey E; Saphire, Erica Ollmann (2009). «Ebolavirus glycoprotein structure and mechanism of entry». Future Virology (em inglês). 4 (6): 621–635. ISSN 1746-0794. PMC 2829775 . PMID 20198110. doi:10.2217/fvl.09.56
- ↑ Swetha, Rayapadi G.; Ramaiah, Sudha; Anbarasu, Anand; Sekar, Kanagaraj (2016). «Ebolavirus Database: Gene and Protein Information Resource for Ebolaviruses». Advances in Bioinformatics (em inglês). 2016. 1673284 páginas. ISSN 1687-8027. PMC 4848411 . PMID 27190508. doi:10.1155/2016/1673284
- ↑ a b c Klenk, H.-D.; Feldmann, H., eds. (2004). Ebola and Marburg Viruses: Molecular and Cellular Biology (em inglês). [S.l.]: Horizon Bioscience. ISBN 978-1904933496
- ↑ Hillman, H. (1991). The Case for New Paradigms in Cell Biology and in Neurobiology (em inglês). [S.l.]: Edwin Mellen Press
- ↑ Zaire ebolavirus isolate H.sapiens-wt/GIN/2014/Makona-Kissidougou-C15, complete genome (em inglês), GenBank
- ↑ Taylor D, Leach R, Bruenn J (2010). «Filoviruses are ancient and integrated into mammalian genomes». BMC Evolutionary Biology (em inglês). 10. 193 páginas. PMC 2906475 . PMID 20569424. doi:10.1186/1471-2148-10-193
- ↑ Belyi, V. A.; Levine, A. J.; Skalka, A. M. (2010). Buchmeier, Michael J., ed. «Unexpected Inheritance: Multiple Integrations of Ancient Bornavirus and Ebolavirus/Marburgvirus Sequences in Vertebrate Genomes». PLOS Pathogens (em inglês). 6 (7): e1001030. PMC 2912400 . PMID 20686665. doi:10.1371/journal.ppat.1001030
- ↑ Taylor DJ, Ballinger MJ, Zhan JJ, Hanzly LE, Bruenn JA (2014). «Evidence that ebolaviruses and cuevaviruses have been diverging from marburgviruses since the Miocene». PeerJ (em inglês). 2: e556. PMC 4157239 . PMID 25237605. doi:10.7717/peerj.556
- ↑ a b Richard Preston (27 de outubro de 2014). «The Ebola Wars». The New Yorker (em inglês). New York: Condé Nast. Consultado em 20 de outubro de 2014
- ↑ a b Gire, Stephen K.; et al. (2014). «Genomic surveillance elucidates Ebola virus origin and transmission during the 2014 outbreak». Science (em inglês). 345 (6202): 1369–1372. Bibcode:2014Sci...345.1369G. PMC 4431643 . PMID 25214632. doi:10.1126/science.1259657
- ↑ Check Hayden, Erika (5 de maio de 2015). «Pint-sized DNA sequencer impresses first users». Nature (em inglês). 521 (7550): 15–16. Bibcode:2015Natur.521...15C. ISSN 0028-0836. PMID 25951262. doi:10.1038/521015a
- ↑ Jenkins GM, Rambaut A, Pybus OG, Holmes EC (2002). «Rates of molecular evolution in RNA viruses: A quantitative phylogenetic analysis». Journal of Molecular Evolution (em inglês). 54 (2): 156–165. Bibcode:2002JMolE..54..156J. PMID 11821909. doi:10.1007/s00239-001-0064-3
- ↑ a b c d Carette JE, Raaben M, Wong AC, Herbert AS, Obernosterer G, Mulherkar N, Kuehne AI, Kranzusch PJ, Griffin AM, Ruthel G, Dal Cin P, Dye JM, Whelan SP, Chandran K, Brummelkamp TR (Setembro de 2011). «Ebola virus entry requires the cholesterol transporter Niemann-Pick C1». Nature (em inglês). 477 (7364): 340–343. Bibcode:2011Natur.477..340C. PMC 3175325 . PMID 21866103. doi:10.1038/nature10348
- Amanda Schaffer (16 de janeiro de 2012). «Key Protein May Give Ebola Virus Its Opening». The New York Times (em inglês)
- ↑ a b c Côté M, Misasi J, Ren T, Bruchez A, Lee K, Filone CM, Hensley L, Li Q, Ory D, Chandran K, Cunningham J (Setembro de 2011). «Small molecule inhibitors reveal Niemann-Pick C1 is essential for Ebola virus infection». Nature (em inglês). 477 (7364): 344–348. Bibcode:2011Natur.477..344C. PMC 3230319 . PMID 21866101. doi:10.1038/nature10380
- Amanda Schaffer (16 de janeiro de 2012). «Key Protein May Give Ebola Virus Its Opening». The New York Times (em inglês)
- ↑ Flemming A (Outubro de 2011). «Achilles heel of Ebola viral entry». Nat Rev Drug Discov (em inglês). 10 (10). 731 páginas. PMID 21959282. doi:10.1038/nrd3568
- ↑ Miller EH, Obernosterer G, Raaben M, Herbert AS, Deffieu MS, Krishnan A, Ndungo E, Sandesara RG, Carette JE, Kuehne AI, Ruthel G, Pfeffer SR, Dye JM, Whelan SP, Brummelkamp TR, Chandran K (Março de 2012). «Ebola virus entry requires the host-programmed recognition of an intracellular receptor». EMBO Journal (em inglês). 31 (8): 1947–1960. PMC 3343336 . PMID 22395071. doi:10.1038/emboj.2012.53
- ↑ Kondratowicz AS, Lennemann NJ, Sinn PL, et al. (Maio de 2011). «T-cell immunoglobulin and mucin domain 1 (TIM-1) is a receptor for Zaire Ebolavirus and Lake Victoria Marburgvirus». Proceedings of the National Academy of Sciences of the United States of America (em inglês). 108 (20): 8426–8431. Bibcode:2011PNAS..108.8426K. PMC 3100998 . PMID 21536871. doi:10.1073/pnas.1019030108
- ↑ Biomarker Database. Ebola virus (em inglês). [S.l.]: Korea National Institute of Health. Consultado em 31 de maio de 2009. Cópia arquivada em 22 de abril de 2008
- ↑ Saeed MF, Kolokoltsov AA, Albrecht T, Davey RA (2010). Basler CF, ed. «Cellular Entry of Ebola Virus Involves Uptake by a Macropinocytosis-Like Mechanism and Subsequent Trafficking through Early and Late Endosomes». PLOS Pathogens (em inglês). 6 (9): e1001110. PMC 2940741 . PMID 20862315. doi:10.1371/journal.ppat.1001110
- ↑ Mühlberger, Elke (4 de dezembro de 2016). «Filovirus replication and transcription». Future Virology (em inglês). 2 (2): 205–215. ISSN 1746-0794. PMC 3787895 . PMID 24093048. doi:10.2217/17460794.2.2.205
- ↑ Feldmann, H.; Klenk, H.-D. (1996). Filoviruses. Medical Microbiology (em inglês). [S.l.]: University of Texas Medical Branch at Galveston. ISBN 978-0963117212. Consultado em 4 de dezembro de 2016
- ↑ Lai, Kang Yiu; Ng, Wing Yiu George; Cheng, Fan Fanny (28 de novembro de 2014). «Human Ebola virus infection in West Africa: a review of available therapeutic agents that target different steps of the life cycle of Ebola virus». Infectious Diseases of Poverty (em inglês). 3. 43 páginas. ISSN 2049-9957. PMC 4334593 . PMID 25699183. doi:10.1186/2049-9957-3-43
- ↑ Quammen, David (30 de dezembro de 2014). «Insect-Eating Bat May Be Origin of Ebola Outbreak, New Study Suggests». news.nationalgeographic.com (em inglês). Washington, DC: National Geographic Society. Consultado em 30 de dezembro de 2014
- ↑ a b Feldmann H (Maio de 2014). «Ebola – A Growing Threat?». N. Engl. J. Med. (em inglês). 371 (15): 1375–1378. PMID 24805988. doi:10.1056/NEJMp1405314
- ↑ Ng, S.; Cowling, B. (2014). «Association between temperature, humidity and ebolavirus disease outbreaks in Africa, 1976 to 2014». Eurosurveillance (em inglês). 19 (35). 20892 páginas. PMID 25210981. doi:10.2807/1560-7917.ES2014.19.35.20892
- ↑ «Clinical care for survivors of Ebola virus disease» (PDF) (em inglês). World Health Organization. 2016. Consultado em 4 de dezembro de 2016
- ↑ Isaacson M, Sureau P, Courteille G, Pattyn, SR. «Clinical Aspects of Ebola Virus Disease at the Ngaliema Hospital, Kinshasa, Zaire, 1976» (em inglês). European Network for Diagnostics of "Imported" Viral Diseases (ENIVD). Consultado em 24 de junho de 2014. Cópia arquivada em 4 de agosto de 2014
- ↑ «Death Called a River». www.scripps.edu (em inglês). Consultado em 9 de outubro de 2014
- ↑ name: S. Reardan.; N Engl. J Med. (2014) " The first nine months of the epidemic and projection, Ebola virus disease in west Africa" (em inglês). archive of Ebola Response Team. 511(75.11):520
- ↑ Gina Kolata (30 de outubro de 2014). «Genes Influence How Mice React to Ebola, Study Says in 'Significant Advance'». New York Times (em inglês). Consultado em 30 de outubro de 2014
- ↑ Rasmussen, Angela L.; et al. (30 de outubro de 2014). «Host genetic diversity enables Ebola hemorrhagic fever pathogenesis and resistance». Science (em inglês). 346 (6212): 987–991. Bibcode:2014Sci...346..987R. PMC 4241145 . PMID 25359852. doi:10.1126/science.1259595
- ↑ Henao-Restrepo, Ana Maria; et al. (22 de dezembro de 2016). «Efficacy and effectiveness of an rVSV-vectored vaccine in preventing Ebola virus disease: final results from the Guinea ring vaccination, open-label, cluster-randomised trial (Ebola Ça Suffit!)». The Lancet (em inglês). 389 (10068): 505–518. PMC 5364328 . PMID 28017403. doi:10.1016/S0140-6736(16)32621-6
- ↑ Berlinger, Joshua (22 de dezembro de 2016). «Ebola vaccine gives 100% protection, study finds». CNN. Consultado em 27 de dezembro de 2016
- ↑ «First FDA-approved vaccine for the prevention of Ebola virus disease, marking a critical milestone in public health preparedness and response». U.S. Food and Drug Administration (FDA) (em inglês). 19 de dezembro de 2019. Consultado em 19 de dezembro de 2019. Cópia arquivada em 20 de dezembro de 2019
- ↑ Brown, Rob (18 de julho de 2014). «The virus detective who discovered Ebola». BBC News (em inglês)
- ↑ Johnson KM, Webb PA, Lange JV, Murphy FA (1977). «Isolation and partial characterisation of a new virus causing haemorrhagic fever in Zambia». Lancet (em inglês). 309 (8011): 569–571. PMID 65661. doi:10.1016/s0140-6736(77)92000-1
- ↑ Netesov SV, Feldmann H, Jahrling PB, Klenk HD, Sanchez A (2000). «Family Filoviridae». In: van Regenmortel MHV, Fauquet CM, Bishop DHL, Carstens EB, Estes MK, Lemon SM, Maniloff J, Mayo MA, McGeoch DJ, Pringle CR, Wickner RB. Virus Taxonomy – Seventh Report of the International Committee on Taxonomy of Viruses (em inglês). San Diego: Academic Press. pp. 539–548. ISBN 978-0123702005
- ↑ Pringle, C. R. (1998). «Virus taxonomy – San Diego 1998». Archives of Virology (em inglês). 143 (7): 1449–1459. PMID 9742051. doi:10.1007/s007050050389
- ↑ Feldmann H, Geisbert TW, Jahrling PB, Klenk HD, Netesov SV, Peters CJ, Sanchez A, Swanepoel R, Volchkov VE (2005). «Family Filoviridae». In: Fauquet CM, Mayo MA, Maniloff J, Desselberger U, Ball LA. Virus Taxonomy – Eighth Report of the International Committee on Taxonomy of Viruses (em inglês). San Diego: Elsevier/Academic Press. pp. 645–653. ISBN 978-0123702005
- ↑ Mayo, M. A. (2002). «ICTV at the Paris ICV: results of the plenary session and the binomial ballot». Archives of Virology (em inglês). 147 (11): 2254–2260. doi:10.1007/s007050200052
- ↑ «Replace the species name Lake Victoria marburgvirus with Marburg marburgvirus in the genus Marburgvirus» (em inglês). Consultado em 31 de outubro de 2014. Cópia arquivada em 5 de março de 2016
- ↑ International Committee on Taxonomy of Viruses. «Virus Taxonomy: 2013 Release» (em inglês)
- ↑ Wahl-Jensen V, Kurz SK, Hazelton PR, Schnittler HJ, Stroher U, Burton DR, Feldmann H (2005). «Role of Ebola Virus Secreted Glycoproteins and Virus-Like Particles in Activation of Human Macrophages». Journal of Virology (em inglês). 79 (4): 2413–2419. PMC 546544 . PMID 15681442. doi:10.1128/JVI.79.4.2413-2419.2005
- ↑ Kesel AJ, Huang Z, Murray MG, Prichard MN, Caboni L, Nevin DK, Fayne D, Lloyd DG, Detorio MA, Schinazi RF (2014). «Retinazone inhibits certain blood-borne human viruses including Ebola virus Zaire». Antiviral Chemistry & Chemotherapy (em inglês). 23 (5): 197–215. PMC 7714485 . PMID 23636868. doi:10.3851/IMP2568
- ↑ a b Carroll, S.A. (2012). «Molecular Evolution of Viruses of the Family Filoviridae Based on 97 Whole-Genome Sequences». Journal of Virology (em inglês). 87 (5): 2608–2616. PMC 3571414 . PMID 23255795. doi:10.1128/JVI.03118-12
- ↑ a b Li, Y.H. (2013). «Evolutionary history of Ebola virus». Epidemiology and Infection (em inglês). 142 (6): 1138–1145. PMC 9151191 . PMID 24040779. doi:10.1017/S0950268813002215
- ↑ a b Wittmann TJ, Biek R, Hassanin A, Rouquet P, Reed P, Yaba P, Pourrut X, Real LA, Gonzalez JP, Leroy EM. "Isolates of Zaire ebolavirus from wild apes reveal genetic lineage and recombinants" (em inglês). Proc Natl Acad Sci U S A. 2007 Oct 23;104(43):17123–17127. Epub 2007 Oct 17. "Erratum" in: Proc Natl Acad Sci U S A. 2007 Dec 4;104(49):19656. PubMed
- ↑ a b «Outbreaks Chronology: Ebola Virus Disease». Ebola Hemorrhagic Feve (em inglês). CDC. 2 de agosto de 2017. Consultado em 11 de novembro de 2017
Bibliografia editar
- Klenk, Hans-Dieter; Feldmann, Heinz (2004). Ebola and Marburg Viruses – Molecular and Cellular Biology. Wymondham, Norfolk, UK: Horizon Bioscience. ISBN 978-0-9545232-3-7
Ligações externas editar
- Informação sobre Ebola, sintomas, tratamento, prevenção. Notícias sobre Ebola atualizadas em tempo real. O link esta inativo.
- ICTV Files and Discussions — Discussion forum and file distribution for the International Committee on Taxonomy of Viruses
- Genomic data on Ebola virus isolates and other members of the Filoviridae family
- Ebola: Africa's Bloody Disease