PETase
PETases são uma classe de enzimas esterásicas que catalisam a hidrólise de plástico PET para o monoetil-2-hidroxietiltereftalato (MHET) monomérico. A reação química idealizada é (onde n é o número de monômeros na cadeia polimérica):
| PETase | |
|---|---|
- (politereftalato de etileno)n + H2O → (politereftalato de etileno)n-1 + MHET
A quantidade vestigial do PET decompõe-se em tereftalato de bis-hidroxietila (BHET). As PETases também podem quebrar o plástico PEF (polietileno-2,5-furandicarboxilato), que é um substituto bioderivado de PET. PETases não podem catalisar a hidrólise de poliésteres alifáticos como succinato de polibutileno ou poliácido láctico. [1]
História
editarA primeira enzima PETase foi descoberta em 2016, a partir da bactéria Ideonella sakaiensis 201-F6 encontrada em amostras de lodo coletadas perto de um local de reciclagem de garrafas PET japonesas.[2] Outros tipos de hidrolases degradadoras de PET são conhecidos antes desta descoberta. Enzimas degradantes de poliéster datam desde 1975 (α-quimotripsina)[3] e 1977 (lipase), por exemplo.[4] O plástico PET foi amplamente utilizado na década de 1970 e tem sido sugerido que as PETases em bactérias evoluíram apenas recentemente.
Divisão MHET em I. sakaiensis
editarA MHET é decomposta em I. sakaiensis pela ação da enzima MHETase em ácido tereftálico e etilenoglicol. Estes são inofensivos ao meio ambiente, pois são decompostos ainda mais para produzir dióxido de carbono e água.[5]
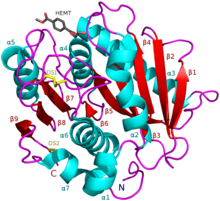
Estrutura
editarEm abril de 2018, havia 13 estruturas cristalinas tridimensionais conhecidas de PETases: 6EQD, 6EQE, 6EQF, 6EQG, 6EQH, 6ANE, 5XJH, 5YNS, 5XFY, 5XFZ, 5XG0, 5XH2 e 5XH3.
Animações e imagens
editar| PET | MHET depois da PETase repartição enzimática do PET |
Animação da separação do PET
para o etilenoglicol e ácido tereftálico |
|---|---|---|
| Resultado final da
separação |
Mais detalhamento | |
Mutações
editarEm 2018, cientistas da Universidade de Portsmouth, com a colaboração do Laboratório Nacional de Energia Renovável do Departamento de Energia dos Estados Unidos, desenvolveram um mutante desse PETase que degrada o PET mais rápido do que aquele em seu estado natural. Neste estudo também foi demonstrado que as PETases podem degradar o polietileno 2,5-furandicarboxilato (PEF).[6]
Referências
editar- ↑ Austin, Harry P.; Allen, Mark D.; Donohoe, Bryon S.; Rorrer, Nicholas A.; Kearns, Fiona L.; Silveira, Rodrigo L.; Pollard, Benjamin C.; Dominick, Graham; Duman, Ramona (17 de abril de 2018). «Characterization and engineering of a plastic-degrading aromatic polyesterase». Proceedings of the National Academy of Sciences (em inglês). ISSN 0027-8424. PMID 29666242. doi:10.1073/pnas.1718804115
- ↑ «Ideonella sakaiensis sp. nov., isolated from a microbial consortium that degrades poly(ethylene terephthalate)». International Journal of Systematic and Evolutionary Microbiology. 66. ISSN 1466-5034. PMID 27045688. doi:10.1099/ijsem.0.001058
- ↑ «Polyester readily hydrolyzable by chymotrypsin». Journal of Polymer Science: Polymer Letters Edition. 13. ISSN 0360-6384. doi:10.1002/pol.1975.130130801
- ↑ «Hydrolysis of polyesters by lipases». Nature. 270. ISSN 0028-0836. PMID 927523. doi:10.1038/270076a0
- ↑ Yoshida, Shosuke; Hiraga, Kazumi; Takehana, Toshihiko; Taniguchi, Ikuo; Yamaji, Hironao; Maeda, Yasuhito; Toyohara, Kiyotsuna; Miyamoto, Kenji; Kimura, Yoshiharu (11 de março de 2016). «A bacterium that degrades and assimilates poly(ethylene terephthalate)». Science (em inglês). 351 (6278): 1196–1199. ISSN 0036-8075. PMID 26965627. doi:10.1126/science.aad6359. Resumo divulgativo (PDF) (30 de março de 2016)
- ↑ «Cientistas desenvolvem por acaso enzima devoradora de plástico». G1