Riboviria
Riboviria é um domínio ou realm de vírus que inclui todos os vírus que usam uma polimerase dependente de RNA homóloga para replicação. Inclui vírus de RNA que codificam uma polimerase de RNA dependente de RNA, bem como vírus de transcrição reversa (com genomas de RNA ou DNA) que codificam uma polimerase de DNA dependente de RNA. A RNA polimerase dependente de RNA (RdRp), também chamada de replicase de RNA, produz RNA ( ácido ribonucléico ) a partir do RNA. A DNA polimerase dependente de RNA (RdDp), também chamada de transcriptase reversa (RT), produz DNA ( ácido desoxirribonucléico ) a partir do RNA. Essas enzimas são essenciais para replicar o genoma viral e transcrever genes virais em RNA mensageiro (mRNA) para tradução de proteínas virais.
| Riboviria | |
|---|---|
| |
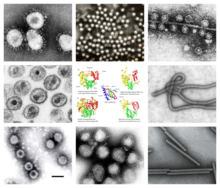
| No sentido dos ponteiros do relógio a partir do canto superior esquerdo: TEM de coronavírus aviário, vírus da poliomielite, bacteriófago Q, Ebolavirus, vírus do mosaico do tabaco, influenzavirus A, rotavirus, VIH-1. Centro: RT e RdRps homólogos com domínio de palma conservado. | |
| Classificação viral | |
| (não classif.): | Virus |
| Realm: | Riboviria |
| Kingdoms | |
A Riboviria foi criada em 2018 para acomodar todos os vírus de RNA que codificam RdRp e foi expandida um ano depois para incluir também retrovírus que codificam RdDp. Esses dois grupos de vírus são atribuídos a dois domínios separados: Orthornavirae para vírus de RNA com codificação RdRp e Pararnavirae para vírus com codificação RdDp, ou seja, todos os vírus de transcrição reversa. Embora o domínio tenha poucos vírus procarióticos, inclui a maioria dos vírus eucarióticos, incluindo a maioria dos vírus humanos, animais e vegetais, no entanto, estudos metagenômicos estão mudando essa perspectiva.
Muitas das doenças virais mais amplamente conhecidas são causadas por vírus em Riboviria, que inclui coronavírus, vírus ebola, HIV, vírus influenza e vírus da raiva. Esses vírus e outros têm se destacado ao longo da história, incluindo o vírus do mosaico do tabaco, que foi o primeiro vírus a ser descoberto. Muitos vírus de transcrição reversa tornam-se notavelmente integrados ao genoma de seu hospedeiro como parte de seu ciclo de replicação. Como resultado disso, estima-se que cerca de 7 a 8% do genoma humano seja originário desses vírus.
Etimologia
editarRiboviria é uma aglutinação de ribo, referindo-se ao ácido ribonucleico, e o sufixo -viria, que é o sufixo usado para domínios de vírus.[1]
Características
editarTodos os membros de Riboviria contêm um gene que codifica uma polimerase dependente de RNA, também chamada de polimerase dirigida por RNA. Existem dois tipos de polimerases dependentes de RNA: a polimerase de RNA dependente de RNA (RdRp), também chamada de replicase de RNA, que sintetiza o RNA a partir do RNA, e a polimerase de DNA dependente de RNA (RdDp), também chamada de transcriptase reversa (RT), que sintetiza ADN a partir de ARN.[2] Em uma partícula viral típica, chamada vírion, a polimerase dependente de RNA está ligada ao genoma viral de alguma maneira e começa a transcrição do genoma viral após entrar na célula. Como parte do ciclo de vida viral, a polimerase dependente de RNA também sintetiza cópias do genoma viral como parte do processo de criação de novos vírus.
Os vírus que se replicam via RdRp pertencem a três grupos no sistema de classificação de Baltimore, todos pertencentes ao domínio Orthornavirae : vírus de RNA de fita simples (ssRNA), que são de sentido positivo (+) ou negativo (-) e duplo- Vírus de RNA encalhado (dsRNA). Os vírus +ssRNA têm genomas que podem atuar funcionalmente como mRNA, e uma fita de sentido negativo também pode ser criada para formar dsRNA a partir da qual o mRNA é transcrito da fita negativa.[3] Os genomas dos vírus -ssRNA e vírus dsRNA atuam como modelos a partir dos quais RdRp cria mRNA.[4][5]
Os vírus que se replicam por transcrição reversa pertencem a dois grupos de Baltimore, ambos pertencentes ao domínio Pararnavirae: vírus de RNA de fita simples (ssRNA-RT), todos pertencentes à ordem Ortervirales, e vírus de DNA de fita dupla (dsDNA-RT ) vírus, que pertencem à família Caulimoviridae, também em Ortervirales, e à família Hepadnaviridae da ordem Blubervirales. Os vírus ssRNA-RT têm seu genoma de sentido positivo transcrito por RdDp para sintetizar uma cadeia de DNA complementar de sentido negativo (-cDNA). A fita +RNA é degradada e posteriormente substituída por RdDp por uma fita +DNA para sintetizar uma cópia linear de dsDNA do genoma viral. Esse genoma é então integrado ao DNA da célula hospedeira.[6]
Para vírus dsDNA-RT, uma fita +RNA pré-genômica é transcrita a partir do DNA circular relaxado (rcDNA), que por sua vez é usado por RdDp para transcrever uma fita -cDNA. A fita +RNA é degradada e substituída de maneira semelhante aos vírus +ssRNA-RT para sintetizar o rcDNA. O genoma rcDNA é posteriormente reparado pelos mecanismos de reparo de DNA da célula hospedeira para sintetizar um genoma de DNA circular fechado covalentemente (cccDNA).[7] O genoma integrado de vírus +ssRNA-RT e o cccDNA de vírus dsDNA-RT são então transcritos em mRNA pela enzima RNA polimerase II da célula hospedeira.[6][7]
O mRNA viral é traduzido pelos ribossomas da célula hospedeira para produzir proteínas virais. Para produzir mais vírus, as polimerases dependentes de RNA viral usam cópias do genoma viral como moldes para replicar o genoma viral. Para vírus +ssRNA, um genoma dsRNA intermediário é criado a partir do qual +ssRNA é sintetizado a partir da fita negativa.[3] Para os vírus -ssRNA, os genomas são sintetizados a partir de cadeias de sentido positivo complementares.[5] os vírus dsRNA replicam seus genomas a partir do mRNA sintetizando uma cadeia de sentido negativo complementar para formar o dsRNA genômico.[4] Para vírus dsDNA-RT, o RNA pré-genômico criado a partir do cccDNA é retrotranscrito em novos genomas dsDNA.[7] Para vírus +ssRNA-RT, o genoma é replicado a partir do genoma integrado.[6] Após a replicação e tradução, o genoma e as proteínas virais são montados em vírions completos, que então deixam a célula hospedeira.
Filogenética
editarAmbos os domínios em Riboviria mostram uma relação com as transcriptases reversas dos íntrons do grupo II que codificam RTs e retrotransposões, que são sequências de DNA autorreplicantes, as quais se autorreplicam por transcrição reversa e se integram a outras partes da mesma molécula de DNA. Os vírus de transcrição reversa, atribuídos a Pararnavirae, parecem ter evoluído de um retrotransposão em uma única ocasião. A origem dos RdRps de Orthornavirae é menos clara devido à falta de informação, que eles se originam de uma transcriptase reversa do íntron bacteriano do grupo II antes do surgimento de eucariotos[2][8][9] ou originaram-se antes do último universal comum ancestral (LUCA) sendo descendentes do antigo mundo do RNA e que eles precederam as transcriptases reversas do retroelemento.[10][11] Um estudo maior (2022), onde novas linhagens (filos) foram descritas, foi a favor da hipótese de que os vírus de RNA descendem do mundo do RNA, sugerindo que os retroelementos se originaram de um ancestral relacionado ao filo Lenarviricota e que os membros de um recém-descoberto Taraviricota linhagem (filo) seriam os ancestrais de todos os vírus de RNA.[12]
Classificação
editarRiboviria contém dois domínios: Orthornavirae e Pararnavirae. Orthornavirae contém vários filos e táxons não atribuídos, enquanto Pararnavirae é monotípico até o nível de classe. Essa taxonomia pode ser visualizada a seguir.[13]
- Domínio: Orthornavirae, que contém todos os vírus de RNA codificadores de RdRp, ou seja, todos os vírus dsRNA, +ssRNA e -ssRNA, geralmente chamados coletivamente de vírus de RNA
- Domínio: Pararnavirae, que contém todos os vírus que codificam RdDp, ou seja, todos os vírus ssRNA-RT e dsDNA-RT, chamados coletivamente de vírus de transcrição reversa
- Filo: Arterviricota
- Classe: Revtraviricetes
- Filo: Arterviricota
Além disso, Riboviria contém duas famílias incertae sedis e quatro gêneros incertae sedis. Informações adicionais sobre eles são necessárias para saber sua colocação exata em táxons superiores. Eles são listados a seguir (- viridae indica família e - vírus indica gênero).[2][13]
Estudos metagenómicos sugeriram a existência de cinco novos filos: Arctiviricota, Taraviricota, Pomiviricota, Paraxenoviricota e Wamoviricota.[12]
Riboviria mescla parcialmente a classificação de Baltimore com a taxonomia de vírus, incluindo os grupos de Baltimore para vírus de RNA e vírus de transcrição reversa no domínio. A classificação de Baltimore é um sistema de classificação usado para vírus com base em sua forma de produção de mRNA, frequentemente usado juntamente com a taxonomia de vírus padrão, baseada na história evolutiva. Todos os membros dos cinco grupos de Baltimore pertencem a Riboviria: Grupo III: vírus dsRNA, Grupo IV: vírus +ssRNA, Grupo V: vírus -ssRNA, Grupo VI: vírus ssRNA-RT e Grupo VII: vírus dsDNA-RT. domínios são o nível mais alto de taxonomia usado para vírus e Riboviria é um dos quatro, sendo os outros três Duplodnaviria, Monodnaviria e Varidnaviria.[8][9][13]
A maioria dos vírus eucarióticos identificados são vírus de RNA e, por essa razão, a maioria dos vírus eucarióticos pertence a Riboviria, incluindo a maioria dos vírus humanos, animais e vegetais. Em contraste, apenas dois grupos de vírus de RNA procarióticos foram identificados: +ssRNA Leviviricetes,[14] que parecem ser alguns dos primeiros vírus de RNA a surgir[8][10] e dsRNA Cystoviridae, que parece estar relacionado a reovírus, que infectam eucariotos.[8] Estudos de amostras megenômicas descobriram novos táxons de vírus de RNA procarióticos, incluindo dois novos filos que infectam apenas procariotos, sugerindo que sua diversidade é maior do que se pensava anteriormente e desafiando a visão tradicional de que os vírus de RNA infectam principalmente eucariotos. Eles também sugerem que as famílias Picobirnaviridae e Partitiviridae previamente associadas a eucariotos também infectam procariotos.[15]
Outros ramos principais de vírus eucarióticos incluem herpesvírus em Duplodnaviria,[16] o domínio Shotokuvirae em Monodnaviria,[17] e muitos vírus em Varidnaviria.[18]
Interações com anfitriões
editarDoença
editarOs vírus em Riboviria estão associados a uma ampla gama de doenças, incluindo muitas das doenças virais mais conhecidas. Notáveis vírus causadores de doenças no domínio incluem: [13]
Os vírus animais em Riboviria incluem os orbivírus, que causam várias doenças em ruminantes e cavalos, incluindo o vírus da língua azul, o vírus da peste equina africana, o vírus da encefalose equina e o vírus da doença hemorrágica epizoótica.[19] O vírus da estomatite vesicular causa doenças em bovinos, equinos e suínos.[20] Os morcegos abrigam muitos vírus, incluindo ebolavírus e henipavírus, que também podem causar doenças em humanos.[21] Da mesma forma, os vírus artrópodes dos gêneros Flavivirus e Phlebovirus são numerosos e frequentemente transmitidos aos humanos.[22][23] Os coronavírus e os vírus influenza causam doenças em vários vertebrados, incluindo morcegos, pássaros e porcos.[24][25] A família Retroviridae contém muitos vírus que causam leucemia, imunodeficiência e outros tipos de câncer e doenças relacionadas ao sistema imunológico em animais.[26][27]
Os vírus de plantas no domínio são numerosos e infectam muitas culturas economicamente importantes. Estima-se que o vírus da murcha manchada do tomate cause mais de 1 bilhão de dólares em danos anualmente, afetando mais de 800 espécies de plantas, incluindo crisântemo, alface, amendoim, pimenta e tomate. O vírus do mosaico do pepino infecta mais de 1.200 espécies de plantas e também causa perdas significativas nas colheitas. O Potato virus Y causa reduções significativas no rendimento e na qualidade da pimenta, batata, tabaco e tomate, e o Plum pox virus é o vírus mais importante entre as culturas de frutas de caroço. O vírus do mosaico bromo, embora não cause perdas econômicas significativas, é encontrado em grande parte do mundo e infecta principalmente gramíneas, incluindo cereais.[13][28]
Endogenização
editarMuitos vírus de transcrição reversa, chamados retrovírus, em Riboviria são capazes de se integrar ao DNA de seu hospedeiro. Esses vírus tornam-se endogenizados como parte de seu ciclo de replicação. Ou seja, o genoma viral é integrado ao genoma do hospedeiro pela enzima retroviral integrase, e o mRNA viral é produzido a partir desse DNA. A endogenização é uma forma de transferência horizontal de genes entre organismos não relacionados, e estima-se que cerca de 7 a 8% do genoma humano consiste em DNA retroviral. A endogenização também pode ser usada para estudar a história evolutiva dos vírus, mostrando um período de tempo aproximado quando um vírus tornou-se endogenizado no genoma do hospedeiro, bem como a taxa de evolução dos vírus desde que a endogenização ocorreu.[29]
Doenças causadas por vírus em Riboviria são conhecidas por grande parte da história registrada, embora sua causa só tenha sido descoberta nos tempos modernos. O vírus do mosaico do tabaco foi descoberto em 1898 e foi o primeiro vírus a ser descoberto.[30] Os vírus transmitidos por artrópodes têm sido centrais no desenvolvimento do controle vetorial, que muitas vezes visa prevenir infecções virais.[31] Na história moderna, numerosos surtos de doenças foram causados por vários membros do domínio, incluindo coronavírus, ebola e influenza.[32] O HIV, especialmente, teve efeitos dramáticos na sociedade, pois causa um declínio acentuado na expectativa de vida e um estigma significativo para as pessoas infectadas.[33][34]
Por muito tempo, a relação entre muitos vírus em Riboviria não pôde ser estabelecida devido à grande quantidade de divergência genética entre os vírus de RNA. Com o desenvolvimento da metagenômica viral, muitos vírus de RNA adicionais foram identificados, ajudando a preencher as lacunas de suas relações.[8] Isso levou ao estabelecimento de Riboviria em 2018 para acomodar todos os vírus de RNA codificadores de RdRp com base na análise filogenética de que eles estavam relacionados.[1]
Um ano depois, todos os vírus de transcrição reversa foram adicionados ao domínio. Os domínios também foram estabelecidos em 2019, separando os dois ramos da polimerase dependente de RNA.[2] Quando o domínio foi fundado, ele incluiu erroneamente duas famílias de viroides, Avsunviroidae e Pospiviroidae, e o gênero Deltavirus, que foram prontamente removidos em 2019 porque usam enzimas de células hospedeiras para replicação.[35]
Ver também
editarReferências
editar- ↑ a b Gorbalenya, Alexander E.; Krupovic, Mart; Siddell, Stuart; Varsani, Arvind; Kuhn, Jens H. (15 de outubro de 2018). «Riboviria: establishing a single taxon that comprises RNA viruses at the basal rank of virus taxonomy» (docx). International Committee on Taxonomy of Viruses (ICTV) (em inglês). Consultado em 9 de março de 2019
- ↑ a b c d Koonin EV, Dolja VV, Krupovic M, Varsani A, Wolf YI, Yutin N, Zerbini M, Kuhn JH (18 de outubro de 2019). «Create a megataxonomic framework, filling all principal taxonomic ranks, for realm Riboviria» (docx). International Committee on Taxonomy of Viruses (ICTV) (em inglês). Consultado em 15 de maio de 2020
- ↑ a b «Positive stranded RNA virus replication». ViralZone. Swiss Institute of Bioinformatics. Consultado em 15 de maio de 2020
- ↑ a b «Double-stranded RNA virus replication». ViralZone. Swiss Institute of Bioinformatics. Consultado em 15 de maio de 2020
- ↑ a b «Negative stranded RNA virus replication». ViralZone. Swiss Institute of Bioinformatics. Consultado em 15 de maio de 2020
- ↑ a b c «ssRNA(RT) replication/transcription». ViralZone. Swiss Institute of Bioinformatics. Consultado em 15 de maio de 2020
- ↑ a b c «dsDNA(RT) replication/transcription». ViralZone. Swiss Institute of Bioinformatics. Consultado em 15 de maio de 2020
- ↑ a b c d e Wolf YI, Kazlauskas D, Iranzo J, Lucia-Sanz A, Kuhn JH, Krupovic M, Dolja VV, Kooning EV (27 de novembro de 2018). «Origins and Evolution of the Global RNA Virome». mBio. 9 (6): e02329-18. PMC 6282212 . PMID 30482837. doi:10.1128/mBio.02329-18
- ↑ a b Krupovic M, Blomberg J, Coffin JM, Dasgupta I, Fan H, Geering AD, Gifford R, Harrach B, Hull R, Johnson W, Kreuze JF, Lindemann D, Llorens C, Lockhart B, Mayer J, Muller E, Olszewski NE, Pappu HR, Pooggin MM, Richert-Poggeler KR, Sabanadzovic S, Sanfacon H, Schoelz JE, Seal S, Stavolone L, Stoye JP, Teycheney PY, Tristem M, Koonin EV, Kuhn JH (15 de junho de 2018). «Ortervirales: New Virus Order Unifying Five Families of Reverse-Transcribing Viruses». J Virol. 92 (12): e00515-18. PMC 5974489 . PMID 29618642. doi:10.1128/JVI.00515-18
- ↑ a b Krupovic M, Dolja VV, Koonin EV (14 de julho de 2020). «The LUCA and its complex virome.» (PDF). Nat Rev Microbiol. 18 (11): 661–670. PMID 32665595. doi:10.1038/s41579-020-0408-x. Consultado em 16 de agosto de 2020
- ↑ Koonin, Eugene V.; Dolja, Valerian V.; Krupovic, Mart (1 de maio de 2015). «Origins and evolution of viruses of eukaryotes: The ultimate modularity». Virology. 479-480: 2–25. PMC 5898234 . PMID 25771806. doi:10.1016/j.virol.2015.02.039
- ↑ a b Zayed, Ahmed A.; et al. (8 de abril de 2022). «Cryptic and abundant marine viruses at the evolutionary origins of Earth's RNA virome». Science. 376 (6589): 156–162. Bibcode:2022Sci...376..156Z. PMID 35389782. doi:10.1126/science.abm5847
- ↑ a b c d e «Virus Taxonomy: 2019 Release». talk.ictvonline.org. International Committee on Taxonomy of Viruses. Consultado em 25 de abril de 2020
- ↑ Callanan J, Stockdale SR, Adriaenssens EM, Kuhn JH, Pallen M, Rumnieks J, Shkoporov A, Draper LA, Ross RP, Hill C (26 de novembro de 2020). «Rename one class (Leviviricetes - formerly Allassoviricetes), rename one order (Norzivirales - formerly Levivirales), create one new order (Timlovirales), and expand the class to a total of six families, 420 genera and 883 species» (docx). International Committee on Taxonomy of Viruses. Consultado em 31 de julho de 2021
- ↑ Neri, Uri; Wolf, Yuri I.; Roux, Simon; Camargo, Antonio Pedro; Lee, Benjamin; Kazlauskas, Darius; Chen, I. Min; Ivanova, Natalia; Allen, Lisa Zeigler (17 de fevereiro de 2022). «A five-fold expansion of the global RNA virome reveals multiple new clades of RNA bacteriophages»: 2022.02.15.480533. doi:10.1101/2022.02.15.480533 – via bioRxiv
- ↑ Koonin EV, Dolja VV, Krupovic M, Varsani A, Wolf YI, Yutin N, Zerbini M, Kuhn JH (18 de outubro de 2019). «Create a megataxonomic framework, filling all principal/primary taxonomic ranks, for dsDNA viruses encoding HK97-type major capsid proteins» (docx). International Committee on Taxonomy of Viruses (em inglês). Consultado em 15 de junho de 2020
- ↑ Koonin EV, Dolja VV, Krupovic M, Varsani A, Wolf YI, Yutin N, Zerbini M, Kuhn JH (18 de outubro de 2019). «Create a megataxonomic framework, filling all principal taxonomic ranks, for ssDNA viruses» (docx). International Committee on Taxonomy of Viruses (em inglês). Consultado em 15 de junho de 2020
- ↑ Koonin EV, Dolja VV, Krupovic M, Varsani A, Wolf YI, Yutin N, Zerbini M, Kuhn JH (18 de outubro de 2019). «Create a megataxonomic framework, filling all principal taxonomic ranks, for DNA viruses encoding vertical jelly roll-type major capsid proteins» (docx). International Committee on Taxonomy of Viruses (em inglês). Consultado em 15 de junho de 2020
- ↑ Maclachlan NJ, Guthrie AJ (dezembro de 2010). «Re-emergence of bluetongue, African horse sickness, and other orbivirus diseases». Vet Res. 41 (6). 35 páginas. PMC 2826768 . PMID 20167199. doi:10.1051/vetres/2010007. Consultado em 15 de agosto de 2020
- ↑ Rozo-Lopez P, Drolet BS, Londono-Renteria B (11 de dezembro de 2018). «Vesicular Stomatitis Virus Transmission: A Comparison of Incriminated Vectors». Insects. 9 (4). 190 páginas. PMC 6315612 . PMID 30544935. doi:10.3390/insects9040190
- ↑ Wang L, Anderson DE (fevereiro de 2019). «Viruses in bats and potential spillover to animals and humans». Curr Opin Virol. 34: 79–89. PMC 7102861 . PMID 30665189. doi:10.1016/j.coviro.2018.12.007
- ↑ Holbrook MR (30 de abril de 2017). «Historical Perspectives on Flavivirus Research». Viruses. 9 (5). 97 páginas. PMC 5454410 . PMID 28468299. doi:10.3390/v9050097
- ↑ Hartman A (junho de 2017). «Rift Valley Fever». Clin Lab Med. 37 (2): 285–301. PMC 5458783 . PMID 28457351. doi:10.1016/j.cll.2017.01.004
- ↑ «Coronaviruses: an overview of their replication and pathogenesis». Coronaviruses. Col: Methods Mol Biol. 1282. [S.l.: s.n.] 2015. pp. 1–23. ISBN 978-1-4939-2437-0. PMC 4369385 . PMID 25720466. doi:10.1007/978-1-4939-2438-7_1
- ↑ Webster RG, Govorkova EA (setembro de 2014). «Continuing challenges in influenza». Ann N Y Acad Sci. 1323 (1): 115–139. Bibcode:2014NYASA1323..115W. PMC 4159436 . PMID 24891213. doi:10.1111/nyas.12462
- ↑ Hartmann K (31 de outubro de 2012). «Clinical aspects of feline retroviruses: a review». Viruses. 4 (11): 2684–2710. PMC 3509668 . PMID 23202500. doi:10.3390/v4112684
- ↑ Payne LN (agosto de 1998). «Retrovirus-induced disease in poultry». Poult Sci. 77 (4): 1204–1212. PMID 9706091. doi:10.1093/ps/77.8.1204
- ↑ Scholthof KB, Adkins S, Czosnek H, Palukaitis P, Jacquot E, Hohn T, Hohn B, Saunders K, Candresse T, Ahlquist P, Hemenway C, Foster GD (dezembro de 2011). «Top 10 plant viruses in molecular plant pathology». Mol Plant Pathol. 12 (9): 938–954. PMC 6640423 . PMID 22017770. doi:10.1111/j.1364-3703.2011.00752.x
- ↑ Aiewsakun P, Katzourakis A (maio de 2015). «Endogenous Viruses: Connecting Recent and Ancient Viral Evolution». Virology. 479-480: 26–37. PMID 25771486. doi:10.1016/j.virol.2015.02.011
- ↑ Harrison BD, Wilson TM (29 de março de 1999). «Milestones in the Research on Tobacco Mosaic Virus». Philos Trans R Soc Lond B Biol Sci. 354 (1383): 521–529. PMC 1692547 . PMID 10212931. doi:10.1098/rstb.1999.0403
- ↑ Wilson AL, Courtenay O, Kelly-Hope LA, Scott TW, Takken W, Torr SJ, Lindsay SW (16 de janeiro de 2020). «The Importance of Vector Control for the Control and Elimination of Vector-Borne Diseases». PLOS Negl Trop Dis. 14 (1): e0007831. PMC 6964823 . PMID 31945061. doi:10.1371/journal.pntd.0007831
- ↑ Norris SL, Sawin VI, Ferri M, Sastre LR, Porgo TV (30 de maio de 2018). «An Evaluation of Emergency Guidelines Issued by the World Health Organization in Response to Four Infectious Disease Outbreaks». PLOS ONE. 13 (5): e0198125. Bibcode:2018PLoSO..1398125N. PMC 5976182 . PMID 29847593. doi:10.1371/journal.pone.0198125
- ↑ Yaghoobi H, Ahmadinia H, Shabani Z, Vazirinejad R, Safari R, Shahizadeh R, Zolfizadeh F, Rezaeian M (31 de dezembro de 2017). «Life Expectancy and Years of Life Lost in HIV Patients Under the Care of BandarAbbas Behavioral Disorders Counseling Center». Nepal J Epidemiol. 7 (4): 702–712. PMC 6204067 . PMID 30510838. doi:10.3126/nje.v7i4.20627
- ↑ Cernasev A, Larson WL, Peden-McAlpine C, Rockwood T, Ranelli PL, Okoro O, Schommer JC (30 de maio de 2020). «"Stigma and HIV Are Like Brother and Sister!": The Experience of African-Born Persons Living With HIV in the US». Pharmacy. 8 (2): E92. PMC 7357078 . PMID 32486263. doi:10.3390/pharmacy8020092
- ↑ Gorbalenya AE, Krupovic M, Siddell S, Varsani A, Kuhn JH (julho de 2019). «Correction of an administrative error resulting in an incorrect taxonomy of the realm Riboviria» (docx). International Committee on Taxonomy of Viruses (ICTV) (em inglês). Consultado em 15 de maio de 2020
Leitura adicional
editar- Ward, C. W. (1993). «Progress towards a higher taxonomy of viruses». Research in Virology. 144 (6): 419–53. PMC 7135741 . PMID 8140287. doi:10.1016/S0923-2516(06)80059-2